[解析事例] Steered MDによるポリペプチドの自由エネルギー変化の解析
- 全原子MD
- 分子構造・親和性・溶解性
- ライフサイエンス
![]() User Only
User Only
反応座標に沿った自由エネルギーの変化を解析
目的と手法
Steered Molecular Dynamics (SMD)では、仮想粒子によって系の構造を強制的に変化させ、その際に仮想粒子にかかる力f から系になされた仕事Wを評価し、構造変化に伴う自由エネルギー変化ΔFを計算することができます。
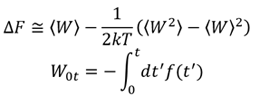
この方法でポリペプチドの末端間距離を反応座標として、伸長状態からヘリックス状態に構造変化する際の自由エネルギー変化を計算しました。図1に示すように、ポリペプチドとしてアラニン10量体をモデリングし、GAFF力場を設定しました。N末端を固定し、C末端に仮想粒子を結合し、末端間距離を変化させるSMD計算を行いました。仕事Wのアンサンブル平均は10個のトラジェクトリから計算しました。仮想粒子の速さvは0.1m/sと1.0m/sで計算を行いました。
 図1 計算に用いたモデル(アラニン10量体)。 N末端を固定しC末端を動かす。
図1 計算に用いたモデル(アラニン10量体)。 N末端を固定しC末端を動かす。
解析結果
計算された自由エネルギーの変化を末端間距離に対してプロットしたものを図2に示します。
図の左にはv=0.1m/sで計算した、伸長状態から縮まる過程(contracting)とヘリックスから伸びていく過程(stretching)の自由エネルギー変化を示しました。末端間距離が長い領域でずれが見られますが、両過程でほぼ同等の結果が得られました。理想的には、過程に依らず同じ自由エネルギー差が得られることが期待されます。
図の右には仮想粒子の移動速度について伸長過程の自由エネルギー変化を比較しており、v=0.1m/sで計算した場合の方が自由エネルギー変化が小さくなりました。
計算された自由エネルギー差は参考文献[1]の結果よりも大きなものになっていますが、これは主に力場の違いによるものと考えられます。
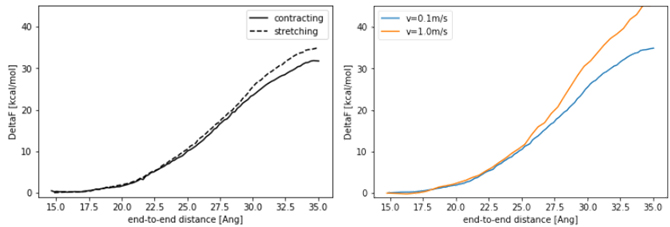 図2 (左) ポリペプチドの伸長状態-ヘリックス状態の自由エネルギー変化。(右) 仮想粒子速度を変えた場合の自由エネルギー変化。
図2 (左) ポリペプチドの伸長状態-ヘリックス状態の自由エネルギー変化。(右) 仮想粒子速度を変えた場合の自由エネルギー変化。
- *参考文献
-
- [1] S. Park, et al., “Free Energy Calculation from Steered Molecular Dynamics Simulations Using Jarzynski ’ s Equality,” J. Chem. Phys., 119, 3553, (2003).
事例一覧
-
- 機械学習ポテンシャルを用いた格子熱伝導率計算
- Martini3モデルによるナフィオン膜の計算
- 機械学習によるσプロファイルを記述子とした物性推算
- 液膜蒸発乾燥シミュレーション
- タイヤの耐摩耗性の向上
- COSMO法による物性推算
- VSOP-PSによる繊維配向材のシミュレーション
- 粗視化モデルによる脂質膜の解析
- 機械学習による沸点、屈折率、比誘電率の推算
- ナノトライボロジー(アブレシブ摩耗、ナノ加工)
- コバルト酸リチウムの基底状態と弾性率の解析(SIESTA事例ページへ)
- MD-GANによる固体電池内のLiイオン拡散解析
- リバースマッピングによるアモルファス構造の作成
- 電池電極の成形プロセス(カレンダリング)における圧力と空隙率の計算
- 粗視化分子動力学を用いた複屈折の解析
- 高分子膜の相分離プロセスシミュレーション
- 機械学習によるχパラメータの推定
- 水への溶解性評価
- フィラー樹脂複合材料の熱伝導率計算
- VSOP-PSによる繊維構造への樹脂含浸プロセス計算
- mol-inferを用いたQSPRの逆解析
- FMO-DPDを用いた高分子電解質の相分離構造
- FMO-DPDを用いた脂質膜とベシクルの形成
- カルサイト(方解石)の複屈折と光吸収(SIESTA事例ページへ)
- リチウムイオン電池のシミュレーション(SIESTA事例ページへ)
- 懸濁液の粘度の評価
- 量子補正を適用した固体の定積比熱の評価
- MD-GANを用いた長時間の分子運動の予測
- ポリマーの誘電緩和
- 金属錯体の吸収スペクトル(SIESTA事例ページへ)
- ゼオライトとガス分子の相互作用(SIESTA事例ページへ)
- 蒸着膜のシミュレーション
- 機械学習QSPRとマテリアルズ・インフォマティクス
- シミュレーション結果を用いた粘弾性マスターカーブの作成
- DPDを用いた粘弾性のシミュレーション
- 表面の再構成(SIESTA事例ページへ)
- フォノン分散を利用した剛性マトリクスの算出(SIESTA事例ページへ)
- Steered MDによるポリペプチドの自由エネルギー変化の解析
- 金属の電子比熱解析(SIESTA事例ページへ)
- 格子比熱の解析(SIESTA事例ページへ)
- J-OCTAによる蓄熱材の評価
- MDおよびMO/DFTを用いた比誘電率の評価
- 溶解度係数の算出
- 機械特性評価(SIESTA事例ページへ)
- 結晶の熱膨張(SIESTA事例ページへ)
- ゼオライトへのガス吸着
- GHz周波数領域における水の誘電分散(2)
- 反応のエネルギー変化(SIESTA事例ページへ)
- GHz周波数領域における水の誘電分散(1)
- 固体表面への分子の吸着エネルギー(SIESTA事例ページへ)
- 活性化エネルギーを用いたモンテカルロ判定によるエポキシ樹脂の架橋反応
- 樹脂 複屈折のためのマルチスケールシミュレーション
- 顔料の表面エネルギー(SIESTA事例ページへ)
- スラリー塗工プロセス
- 金属表面と分子の相互作用
- フィラー充填ゴムの繰り返し伸長
- 界面特性を考慮したCFRTPの破壊挙動の解析(Digimat事例ページへ)
- グラフェンシート添加によるCFRTPのマトリクス熱伝導特性改質(Digimat事例ページへ)
- 非平衡MDによる熱伝導率の計算
- ゴム材料のレオロジーシミュレーション
- 架橋フェノール樹脂の物性評価
- エポキシ樹脂架橋反応のシミュレーションからのガラス転移温度評価
- 電池用電解液の計算
- カーボンナノチューブの分散構造と非線形構造解析
- 粗視化ポテンシャルの評価
- DPDによる溶媒蒸発シミュレーション
- ガラス状態ポリマーの一軸伸長とクレーズ形成
- 高分子-固体界面の摩擦
- MDによる粘度の評価
- 相図からのχパラメータ推算
- 高分子-固体界面の剥離
- 気体の溶解係数と自由体積
- QSPRによる物性値の推算
- レオロジー特性
- 熱硬化性樹脂の架橋構造
- MDによる比誘電率の評価
- 複合材料の非線形力学特性(LS-DYNAとの連携)
- DPDによる界面張力の評価
- MDによる溶解度パラメータの評価
- DPDによる液滴のずり変形
- MDによる界面張力の評価
- 体積弾性率評価
- 界面活性剤の影響評価
- 燃料電池の高分子電解質膜解析
- 一軸伸張解析
- 光学特性評価
- ガラス転移温度評価
- ガス拡散解析
- ガス透過解析
- 架橋構造材料の特性評価
- コンポジット材料の特性解析
- ※記載されている製品およびサービスの名称は、それぞれの所有者の商標または登録商標です。
- ※ お問い合わせページへアクセスできない場合
-
以下のアドレス宛にメールでお問い合わせください






